产品简介 Product Introduction
指纹图谱是由英国遗传学家Jeffreys于1986年提出,是一种具有快速、准确特点的鉴定品种的有力工具,已广泛应用于很多物种的品种资源多样性评估。DNA指纹图谱直接反映生物个体在DNA水平上的差异,具有高度的个体特异性和环境稳定性,如同人类的指纹作为身份标识一样,植物的DNA指纹图谱为其贴上了“分子身份证”。构建基于SNP标记技术的植物品种DNA指纹图谱,可以克服以往依赖性状特征鉴定品种的局限,在品种鉴定与知识产权的保护、品种纯度、品种亲缘关系和分类研究中具有重要意义。
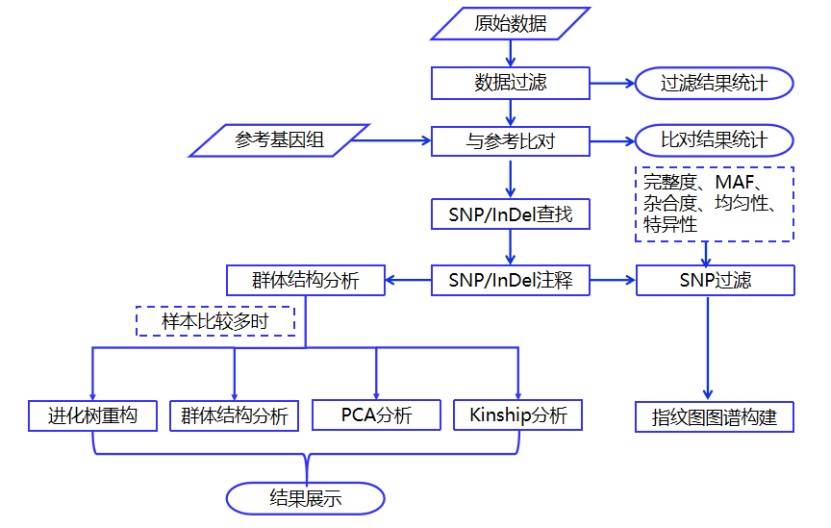
分析流程 Analysis Process
测序策略 Sequencing Strategy
技术选择 | 平台 | 测序深度/数量量 | 备注 |
重测序 | Illumina/BGI | 10-20X | |
简化基因组测序 | Illumina/BGI | 10-20万标记 | 标记越多准确性越高 |
分析内容 Analysis Content
1. 测序数据质控
2. 与参考基因组比对
3. SNP检测与注释
4. InDel检测与注释
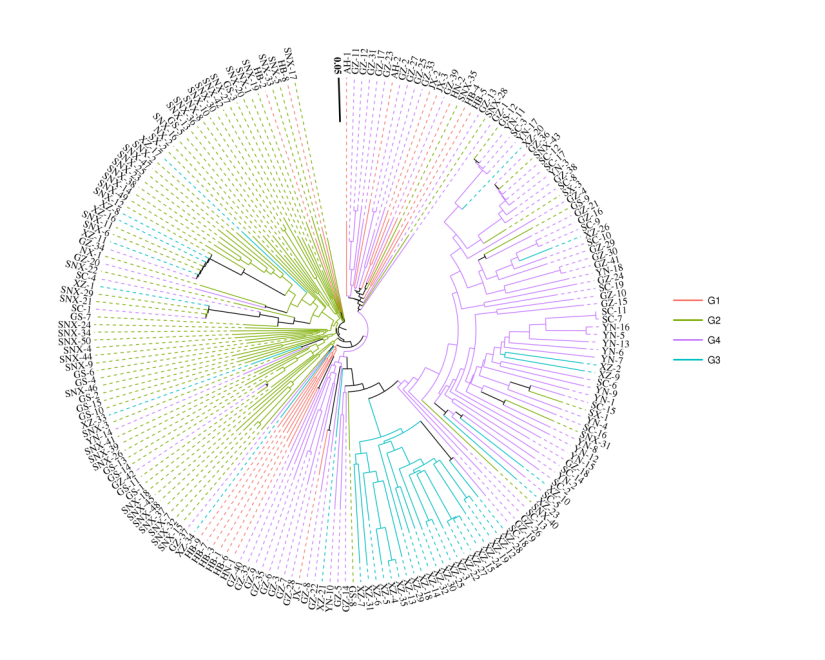
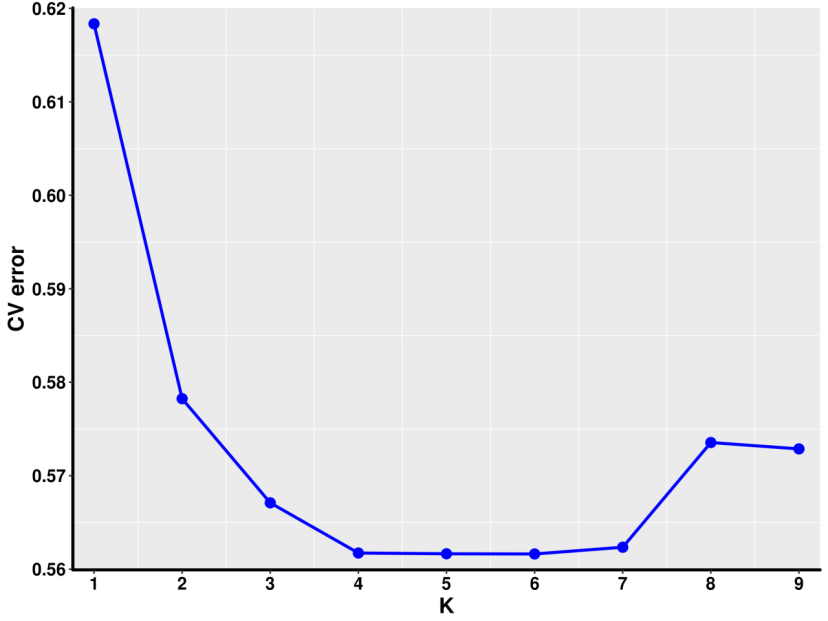
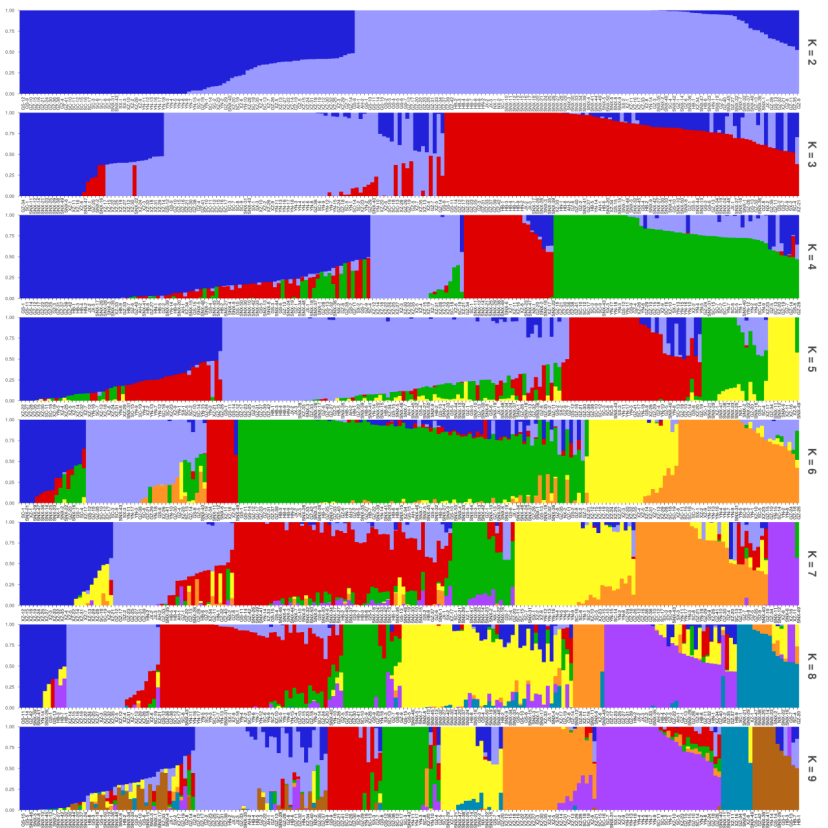
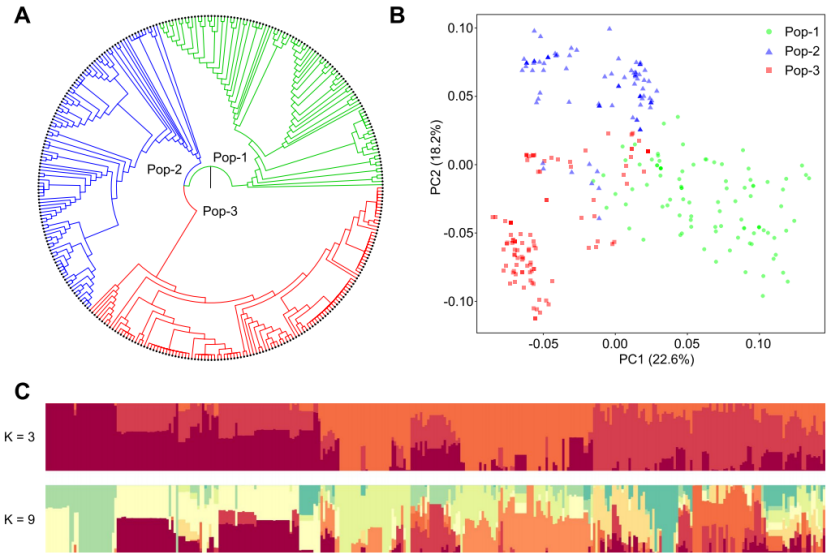
5. 基于SNP的群体结构分析(样本数目>10个)
1) 群体进化树构建(NJ树)
2) 群体结构分析(Admixture)
3) 群体主成分分析(PCA)
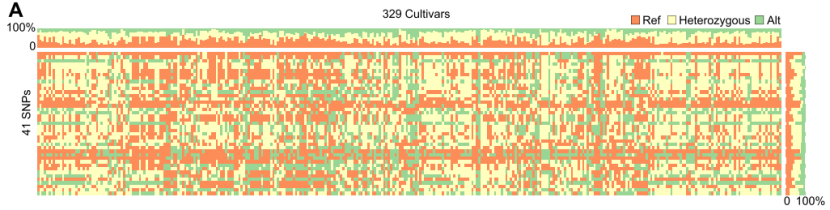
6. SNP过滤(根据snp完整度、MAF、杂合信息等过滤)
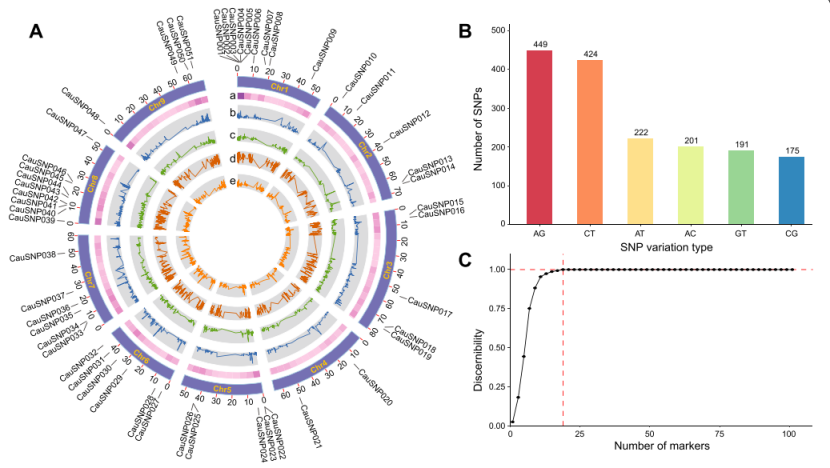
7. 特有SNP筛选
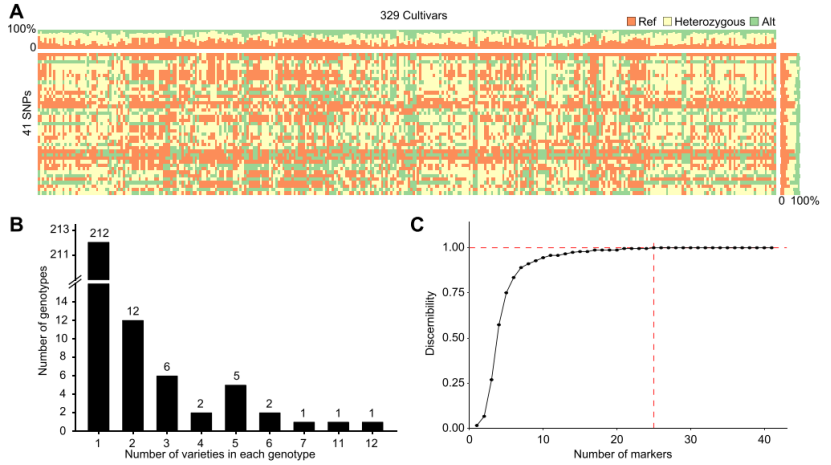
8. 指纹图谱开发
© 2025 天津启源生物科技发展有限公司 版权所有
 津ICP备2025029719号-1
津ICP备2025029719号-1
微信

业务咨询
18102110725
电话
18102110725
返回顶部